Nanorotor und -greifer bringen DNA-Origami einen großen Schritt weiter
Bewegung und Präzision: Die neuesten Nanomaschinen aus dem Labor Dietz
2016-03-04 – Nachrichten aus dem Physik-Department
Inspiriert von Nanomaschinen in der Natur – beispielsweise das Enzym ATP-Synthase und die motorgetriebenen Flagellen von Bakterien – bauen die Physiker um Hendrik Dietz ihr eigenes Design- und Konstruktionsrepertoire immer weiter aus. Das Team entwickelt systematisch Regeln und Prozesse, um auf der Grundlage von DNA-Origami mit immer größerer Flexibilität und immer genauerer Steuerung sich selbst montierende Strukturen zu erzeugen. Seit dem anfänglichen Zusammenfügen komplementärer DNA-Basenpaare hat sich der Werkzeugkasten der Forscher stetig in Richtung modularer Aufbauten und der Programmierung übergeordneter Bausteine fortentwickelt. Heute bauen sie ihre Konstruktionen mit einer Vielzahl gegengleich geformter, ineinandergreifender Bausteine.
Im Zuge dieser Entwicklung haben die Forscher auch ihre Verifizierungsmethoden verfeinert. Sie dienen zum Beispiel dem Nachweis, dass eine bestimmte Lösung mit Nanopartikeln wirklich voller Kopien des ursprünglich gestalteten Objekts ist. Das Objekt könnte zum Beispiel ein schaltbares Getriebe, ein künstlicher Membrankanal, ein willkürlich gestaltetes, komplexes Testobjekt, ein sich öffnendes und schließendes „Nanobuch“ oder ein Roboter mit beweglichen Armen sein.
Zwei Neuzugänge im Zoo
Die beiden Neuzugänge im Zoo der DNA-Origami Objekte des Labors, zwei winzige 3D-Maschinen mit beweglichen Teilen, demonstrieren nun neue Einsatzmöglichkeiten. (Video-Animation auf EurekAlert, 1‘23 Min.)
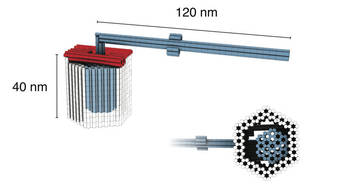
Die Doktoranden Philip Ketterer und Elena Willner bauten zusammen mit Dietz einen Drehmechanismus aus drei mehrlagigen DNA-Bausteinen: Einen Rotor aus einem etwa 32 Nanometer langen Körper mit einem längeren, hebel-ähnlichen Arm und zwei Klemmelemente, die „zusammenklappen“ und so ein Achslager formen. Die beiden Klemmen fügen sich sehr fest zusammen und lassen nur zwei Nanometer Spiel um die Achse, so dass sich der Rotor drehen kann, aber nicht eiert. In einer Version wird der Arm frei zwischen zwei willkürlich gewählten Endpunkten rotieren, in einer anderen Variante wird er in bestimmten Positionen, sogenannten „docking sites“ verweilen. Bis heute ist dies die komplexeste rotierende Struktur, die mit DNA-Origami realisiert wurde.
Der Rotor hat natürlich keinen Motor. Sein Antrieb entstammt der Brownschen Molekularbewegung, also der statistischen Bewegung von Teilchen in einer Lösung. Allerdings eröffnen die Forscher, indem sie die Machbarkeit einer solchen Maschine demonstrieren, den Weg für aktive Varianten, die chemisch oder thermisch gesteuert und angetrieben werden. “Es ist, als hätten wir einen Verbrennungsmotor gebaut,” erklärt Dietz, “und nun stünden als nächstes die Zündkerzen und der Treibstoff an.”
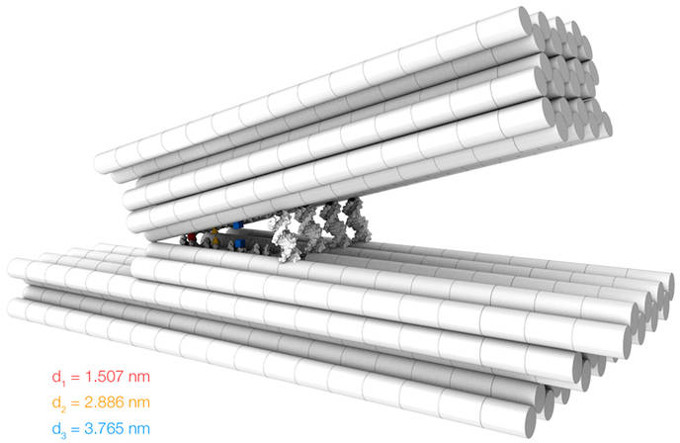
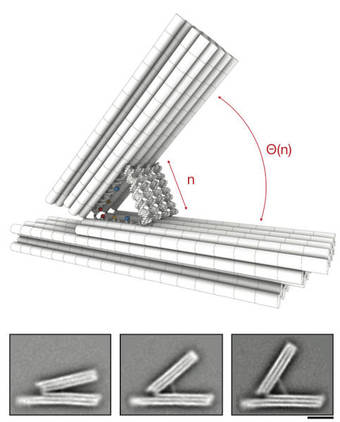
In einem weiteren Projekt realisierte Dietz mit dem Doktorand Jonas Funke eine Maschine mit Klappmechanismus, die sich von der Größe her sich dazu eignet, einzelne Moleküle mit atomarer Präzision zu manipulieren. Der Winkel zwischen den zwei Greifelementen wird mit DNA-Helices gesteuert. Experimente mit diesem Positionier-Gerät auf der Grundlage von DNA-Origami zeigten, dass es Moleküle präzise platzieren könnte, indem es den Abstand zwischen ihnen in Schritten anpasst, die so klein sind wie der Radius eines Wasserstoffatoms. Die Bedeutung dieser Arbeit liegt darin, dass sie dem Trend des Forschungsfelds entgegensteht, mit DNA-Origami immer größere Konstruktion zu bauen, ohne dabei unbedingt die erreichbare Präzision weiter zu auszureizen. Außerdem deuten die Ergebnisse einen von mehreren Wegen an, wie DNA-Nanomaschinen eines Tages für die Steuerung chemischer Reaktionen genutzt werden könnten.
“Zum einen vertrauen wir nun wirklich der Platzierungsgenauigkeit innerhalb unserer Strukturen, weil wir tatsächlich zwei Moleküle platziert und ihren Abstand dabei mit atomarer Präzision kontrolliert haben,” sagt Dietz. “Zum anderen haben wir jetzt einen ersten Prototyp für einen Drehmechanismus. Aus unseren Messungen schließen wir, dass seine Beweglichkeit bis zu 50.000 Umdrehungen pro Minute zuließe, wenn jede durch einen thermischen Stoß ausgelöste Winkeländerung in dieselbe Richtung ginge. In der nächsten Generation werden wir die Platzierungsgenauigkeit ausnutzen, um chemische Reaktionen an die Bewegungen eines Rotors zu koppeln. So könnte aus dem Rotor ein Motor werden. Dieser Motor könnte dann für vielerlei Zwecke dienen, beispielsweise um als Propeller einen winzigen, nanometerkleinen Arzneistoff-Träger (Drug Delivery Vehicle) anzutreiben, um Moleküle zum Separieren durch Membranen zu pumpen, oder um Moleküle in bestimmte Räume, sogenannte Kompartimente zu verpacken.”
Die Arbeiten wurden unterstützt vom Europäischen Forschungsrat (ERC Starting Grant für H.D.), von der Deutschen Forschungsgemeinschaft im Rahmen der Exzellenzcluster CIPSM und NIM, des Sonderforschungsbereichs (SFB) 863 und der Deutsch-Israelischen Projektkooperation, sowie von der TUM International Graduate School for Science and Engineering.
Veröffentlichungen
- Nanoscale rotary apparatus formed from tight-fitting 3D DNA componentsPhilip Ketterer, Elena M. Willner and Hendrik Dietz
- Placing molecules with Bohr radius resolution using DNA origamiJonas J. Funke, Hendrik Dietz
Video (frei zur Verwendung)
Verwandte Meldungen
- Winkender Nanoroboter signalisiert neue Flexibilität im “DNA-Origami” – 2015-03-27
- Biophysiker Hendrik Dietz (36) erhält deutsche Spitzenauszeichnung – 2014-12-10
- Schnelles Falten von DNA in Nanostrukturen bei konstanter Temperatur – 2012-12-14
- Cryo-Elektronenmikroskopie erlaubt tiefe Einblicke in 3D-DNA-Objekte – 2012-12-06
- Eine synthetische Nanopore imitiert den Transport von Substanzen durch Zellmembranen – 2012-11-19
Links
Kontakt
- Prof. Hendrik Dietz
- Technische Universität MünchenJames-Franck-Str. 185748 GarchingTel.: +49 89 289-11615E-Mail: dietz@tum.de