Biophysik
 Dietz Lab. Bild: TUM/Eckert. In der Biophysik werden einerseits die elementaren molekularen Komponenten einer Zelle als auch das Zusammenwirken selbiger auf der Systemebene untersucht. Dafür werden neue experimentelle Messmethoden und theoretische Beschreibungsweisen entwickelt und angewendet. Das Ziel der Forschung ist ein möglichst umfassendes Verständnis der fundamentalen Mechanismen, die letztendlich Leben ermöglichen oder aber auch Krankheiten verursachen. Diese Erkenntnisse können letztlich beispielsweise zur Diagnose und Heilung von Krankheiten beitragen, oder aber auch neue biologisch-inspirierte Technologien wie z.B künstliche Photosynthese oder auch selbstorganisierte Fabrikation ermöglichen. Die Komplexität der lebenden Materie bietet dabei die faszinierende Gelegenheit, vollkommen neue physikalische Gesetzmäßigkeiten fern vom chemischen oder thermodynamischen Gleichgewicht zu entdecken. Die Größe unseres Forschungsbereiches ermöglicht es nahezu alle Aspekte dieses modernen und hochgradig interdisziplinären Forschungsgebietes abzudecken.
Dietz Lab. Bild: TUM/Eckert. In der Biophysik werden einerseits die elementaren molekularen Komponenten einer Zelle als auch das Zusammenwirken selbiger auf der Systemebene untersucht. Dafür werden neue experimentelle Messmethoden und theoretische Beschreibungsweisen entwickelt und angewendet. Das Ziel der Forschung ist ein möglichst umfassendes Verständnis der fundamentalen Mechanismen, die letztendlich Leben ermöglichen oder aber auch Krankheiten verursachen. Diese Erkenntnisse können letztlich beispielsweise zur Diagnose und Heilung von Krankheiten beitragen, oder aber auch neue biologisch-inspirierte Technologien wie z.B künstliche Photosynthese oder auch selbstorganisierte Fabrikation ermöglichen. Die Komplexität der lebenden Materie bietet dabei die faszinierende Gelegenheit, vollkommen neue physikalische Gesetzmäßigkeiten fern vom chemischen oder thermodynamischen Gleichgewicht zu entdecken. Die Größe unseres Forschungsbereiches ermöglicht es nahezu alle Aspekte dieses modernen und hochgradig interdisziplinären Forschungsgebietes abzudecken.
Sprecher des Forschungsbereiches ist Matthias Rief.
Molekulare Biophysik
Arbeitsgruppen: Rief - Simmel - Hugel - Dietz
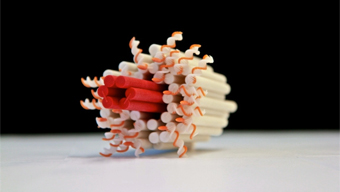 Mit der DNA-Nanotechnologie können neuartige Moleküle gebaut werden, hier ist ein künstlicher Membran-Kanal dargestellt. Bild: TUM.PH/Dietz Lab. In der Evolution des Lebens sind faszinierende molekulare Maschinen entstanden, die zum überwiegenden Teil aus Proteinen aufgebaut sind. Diese molekularen Maschinen sind an allen zellulären Prozessen maßgeblich beteiligt. Die Bandbreite ihrer biologischen Funktion reicht von einfachen Schaltern über molekulare Motoren, die Ladung entlang molekularer Schienen transportieren, bis hin zu komplexen chemischen Syntheseeinheiten, in denen beispielsweise die Erbinformation repliziert wird.
Mit der DNA-Nanotechnologie können neuartige Moleküle gebaut werden, hier ist ein künstlicher Membran-Kanal dargestellt. Bild: TUM.PH/Dietz Lab. In der Evolution des Lebens sind faszinierende molekulare Maschinen entstanden, die zum überwiegenden Teil aus Proteinen aufgebaut sind. Diese molekularen Maschinen sind an allen zellulären Prozessen maßgeblich beteiligt. Die Bandbreite ihrer biologischen Funktion reicht von einfachen Schaltern über molekulare Motoren, die Ladung entlang molekularer Schienen transportieren, bis hin zu komplexen chemischen Syntheseeinheiten, in denen beispielsweise die Erbinformation repliziert wird.
 Mit optischen Pinzetten wird die Mechanik von einzelnen Molekülen bestimmt. Bild: TUM.PH/Dietz Lab. Ultrasensitive Messmethoden ermöglichen einzigartige Einblicke in die Funktionsweise dieser faszinierenden molekularen Maschinen. Deshalb arbeiten wir konzentriert an der Weiterentwicklung und Anwendung von mechanischen und optischen Einzelmolekülmethoden, sowie hochauflösenden Mikroskopiemethoden. Durch die Kombination von biochemischen und biophysikalischen Methoden ist es auch möglich neuartige Nanobauteile herzustellen, um einerseits einzelne Moleküle gezielter zu manipulieren oder aber auch z.B natürliche Enzyme künstlich nachzubauen.
Mit optischen Pinzetten wird die Mechanik von einzelnen Molekülen bestimmt. Bild: TUM.PH/Dietz Lab. Ultrasensitive Messmethoden ermöglichen einzigartige Einblicke in die Funktionsweise dieser faszinierenden molekularen Maschinen. Deshalb arbeiten wir konzentriert an der Weiterentwicklung und Anwendung von mechanischen und optischen Einzelmolekülmethoden, sowie hochauflösenden Mikroskopiemethoden. Durch die Kombination von biochemischen und biophysikalischen Methoden ist es auch möglich neuartige Nanobauteile herzustellen, um einerseits einzelne Moleküle gezielter zu manipulieren oder aber auch z.B natürliche Enzyme künstlich nachzubauen.
Zelluläre Biophysik
Arbeitsgruppen: Alim -Bausch - Simmel
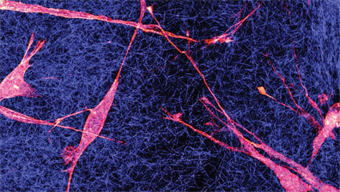
 Musterbildung in einem rekonstruierten Zytoskelettsystem. Bild: PH.TUM/Bausch Lab. Wie kann aus einzelnen Molekülen so etwas faszinierenden anderes wie Leben entstehen? Alle lebenden Organismen gehorchen physikalischen Gesetzen – auch wenn die Vielfalt und Möglichkeiten ersteinmal qualitativ eine ganz andere Dimension darstellen, als wir von der toten Materie her kennen. Wie können wir so komplizierte Prozesse wie Zellteilung oder Embryonalentwicklung verstehen? Während in der molekularen Biologie oder Biochemie die Identifizierung der molekularen Ursachen im Vordergrund stehen, kann es nur mit physikalischen Konzepten und Methoden gelingen, die übergeordneten Prinzipien zu verstehen. Es ist die Aufgabe der Biophysik ein Verständnis in die Vielfalt der 30.000 identifizierten Proteine mit all ihren Interaktionen zu bringen. Die rasanten Entwicklungen von vielen physikalischen Mess- und Analyse Methoden (insbesondere der optischen Messtechniken, optische Bildverarbeitung, Mikromechanik, Mikrostrukturierung oder Mikrofluidik) führt zu sehr aufregenden neuen Möglichkeiten, dem was Leben möglich macht, auf die Spur zu kommen.
Musterbildung in einem rekonstruierten Zytoskelettsystem. Bild: PH.TUM/Bausch Lab. Wie kann aus einzelnen Molekülen so etwas faszinierenden anderes wie Leben entstehen? Alle lebenden Organismen gehorchen physikalischen Gesetzen – auch wenn die Vielfalt und Möglichkeiten ersteinmal qualitativ eine ganz andere Dimension darstellen, als wir von der toten Materie her kennen. Wie können wir so komplizierte Prozesse wie Zellteilung oder Embryonalentwicklung verstehen? Während in der molekularen Biologie oder Biochemie die Identifizierung der molekularen Ursachen im Vordergrund stehen, kann es nur mit physikalischen Konzepten und Methoden gelingen, die übergeordneten Prinzipien zu verstehen. Es ist die Aufgabe der Biophysik ein Verständnis in die Vielfalt der 30.000 identifizierten Proteine mit all ihren Interaktionen zu bringen. Die rasanten Entwicklungen von vielen physikalischen Mess- und Analyse Methoden (insbesondere der optischen Messtechniken, optische Bildverarbeitung, Mikromechanik, Mikrostrukturierung oder Mikrofluidik) führt zu sehr aufregenden neuen Möglichkeiten, dem was Leben möglich macht, auf die Spur zu kommen.
Theoretische Biophysik
Arbeitsgruppen: Alim -Gerland - Zacharias - Emeritus van Hemmen
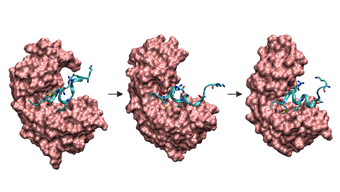 Simulation der gekoppelten Faltung und Bindung eines Peptids an ein Protein. In der theoretischen Biophysik untersucht man biologische Systeme und versucht sie mit Hilfe theoretischer Konzepte aus der Physik zu erklären. Die Eigenschaften biologischer Systeme beruhen auf dem komplizierten Wechselspiel und der Dynamik einer Vielzahl molekularer Komponenten. Molekulare Simulationsmethoden erlauben die Untersuchung der Struktur und Dynamik biologischer Moleküle. Beispielsweise nehmen Biomoleküle wie Proteine, definierte räumliche Strukturen ein, die ihre Funktion bestimmen. Solche Strukturbildungs- oder Faltungsvorgänge können durch moderne Simulationsmethoden bei atomarer Auflösung und unter Einbeziehung umgebender Wassermoleküle und Ionen nachvollzogen werden. Dies erlaubt eine Analyse der zu Grunde liegenden strukturbildenden Kräfte und thermodynamischer und kinetischer Parameter. Viele noch ungeklärte Fragen zum Mechanismus der spezifischen Strukturbildung können so durch molekulare Simulationen aufgeklärt werden.
Simulation der gekoppelten Faltung und Bindung eines Peptids an ein Protein. In der theoretischen Biophysik untersucht man biologische Systeme und versucht sie mit Hilfe theoretischer Konzepte aus der Physik zu erklären. Die Eigenschaften biologischer Systeme beruhen auf dem komplizierten Wechselspiel und der Dynamik einer Vielzahl molekularer Komponenten. Molekulare Simulationsmethoden erlauben die Untersuchung der Struktur und Dynamik biologischer Moleküle. Beispielsweise nehmen Biomoleküle wie Proteine, definierte räumliche Strukturen ein, die ihre Funktion bestimmen. Solche Strukturbildungs- oder Faltungsvorgänge können durch moderne Simulationsmethoden bei atomarer Auflösung und unter Einbeziehung umgebender Wassermoleküle und Ionen nachvollzogen werden. Dies erlaubt eine Analyse der zu Grunde liegenden strukturbildenden Kräfte und thermodynamischer und kinetischer Parameter. Viele noch ungeklärte Fragen zum Mechanismus der spezifischen Strukturbildung können so durch molekulare Simulationen aufgeklärt werden.
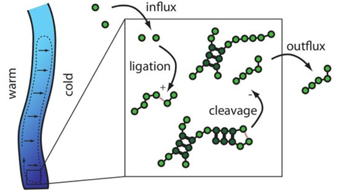 Modell eines thermisch getriebenen Reaktionssystems. Biologische Systeme sind nicht nur auf molekularer Ebene sondern auch in höheren Organisationseinheiten wie Zellorganellen oder ganzen Zellen hochdynamisch. Hier spielen häufig stochastische Prozesse fern vom thermodynamischen Gleichgewicht eine wesentliche Rolle. Das Wechselspiel dieser Vorgänge bestimmt und reguliert zelluläre Prozesse und führt zur Selbstorganisation zellulärer Strukturen. Beispielsweise laufen in Zellen stochastische Prozesse ab, die biochemische Energie verbrauchen und damit eine gerichtete Bewegung erzeugen. Mit Hilfe mathematischer Methoden und Simulationen auf mesoskopischen Skalen können Modelle zur Beschreibung und Erklärung solcher Prozesse erstellt werden. Dabei sind insbesondere die Prinzipien der Organisation und Regulation stochastischer Prozesse in Zellen und deren Entwicklung im Laufe der Evolution noch weitgehend unverstanden und Gegenstand unserer Forschung.
Modell eines thermisch getriebenen Reaktionssystems. Biologische Systeme sind nicht nur auf molekularer Ebene sondern auch in höheren Organisationseinheiten wie Zellorganellen oder ganzen Zellen hochdynamisch. Hier spielen häufig stochastische Prozesse fern vom thermodynamischen Gleichgewicht eine wesentliche Rolle. Das Wechselspiel dieser Vorgänge bestimmt und reguliert zelluläre Prozesse und führt zur Selbstorganisation zellulärer Strukturen. Beispielsweise laufen in Zellen stochastische Prozesse ab, die biochemische Energie verbrauchen und damit eine gerichtete Bewegung erzeugen. Mit Hilfe mathematischer Methoden und Simulationen auf mesoskopischen Skalen können Modelle zur Beschreibung und Erklärung solcher Prozesse erstellt werden. Dabei sind insbesondere die Prinzipien der Organisation und Regulation stochastischer Prozesse in Zellen und deren Entwicklung im Laufe der Evolution noch weitgehend unverstanden und Gegenstand unserer Forschung.
System-Biophysik
Arbeitsgruppen: Bausch - Simmel - Emeritus van Hemmen
 Epithelzellen interagieren über die extrazelluläre Matrix miteinander. Bild: TUM.PH/Bausch Lab. Biologische Systeme sind hochdynamisch und viele ihrer Eigenschaften beruhen auf dem komplizierten Wechselspiel einer Vielzahl molekularer Komponenten oder Organisationseinheiten. Unser Ziel ist es, verschiedenste Aspekte dieser Dynamik besser zu verstehen und auch zu steuern. Auf der mikroskopischen Ebene spielt thermisch getriebenes, stochastisches Verhalten eine wesentliche Rolle. Auch laufen viele Prozesse ab, die biochemische Energie verbrauchen und damit z.B. gerichtete Bewegung erzeugen. Biosysteme sind deshalb fern vom thermodynamischen Gleichgewicht. Ihre komplexe Struktur verursacht oft Abweichungen vom Standardverhalten einfacherer Systeme, was zu spannenden neuen physikalischen Fragestellungen führt. Beispielsweise vollführen passive Teilchen im superdichten Zellinneren eine sogenannte "anomale Diffusionsbewegung". Diese wiederum trägt zum Verständnis vielfältiger molekularer Prozesse wie der Genregulation, zellulärer Verteidigungsmechanismen oder der Zellteilung bei.
Epithelzellen interagieren über die extrazelluläre Matrix miteinander. Bild: TUM.PH/Bausch Lab. Biologische Systeme sind hochdynamisch und viele ihrer Eigenschaften beruhen auf dem komplizierten Wechselspiel einer Vielzahl molekularer Komponenten oder Organisationseinheiten. Unser Ziel ist es, verschiedenste Aspekte dieser Dynamik besser zu verstehen und auch zu steuern. Auf der mikroskopischen Ebene spielt thermisch getriebenes, stochastisches Verhalten eine wesentliche Rolle. Auch laufen viele Prozesse ab, die biochemische Energie verbrauchen und damit z.B. gerichtete Bewegung erzeugen. Biosysteme sind deshalb fern vom thermodynamischen Gleichgewicht. Ihre komplexe Struktur verursacht oft Abweichungen vom Standardverhalten einfacherer Systeme, was zu spannenden neuen physikalischen Fragestellungen führt. Beispielsweise vollführen passive Teilchen im superdichten Zellinneren eine sogenannte "anomale Diffusionsbewegung". Diese wiederum trägt zum Verständnis vielfältiger molekularer Prozesse wie der Genregulation, zellulärer Verteidigungsmechanismen oder der Zellteilung bei.
 Erste Schritte hin zu einer minimalen künstlichen Zellen: Fluoreszenzbild eines Transkriptions-Oszillators in einer Mikroemulsion. Bild: TUM.PH/Simmel Lab. Auf der Ebene neuronaler Zellverbände sind wir an sensorischer Informationsverarbeitung interessiert. Auf Basis verschiedener Inputs von Hör- und Sehorganen, aber auch des Seitenliniensystems von Fischen und Fröschen oder des Infrarotorgans von Schlangen wird im Gehirn eine einheitliche Darstellung der sensorischen Umgebung gebildet, die eine Entscheidungsfindung ermöglicht. Speziell die Dynamik dieser Prozesse steht hierbei im Zentrum unseres Forschungsinteresses.
Erste Schritte hin zu einer minimalen künstlichen Zellen: Fluoreszenzbild eines Transkriptions-Oszillators in einer Mikroemulsion. Bild: TUM.PH/Simmel Lab. Auf der Ebene neuronaler Zellverbände sind wir an sensorischer Informationsverarbeitung interessiert. Auf Basis verschiedener Inputs von Hör- und Sehorganen, aber auch des Seitenliniensystems von Fischen und Fröschen oder des Infrarotorgans von Schlangen wird im Gehirn eine einheitliche Darstellung der sensorischen Umgebung gebildet, die eine Entscheidungsfindung ermöglicht. Speziell die Dynamik dieser Prozesse steht hierbei im Zentrum unseres Forschungsinteresses.
Ein Hauptaspekt des Arbeitsbereiches Dynamik von Biosystemen ist es, die oben beschriebenen dynamischen Prozesse experimentell und theoretisch zu untersuchen, indem sie in synthetischen biologischen Systemen kontrolliert "nachgebaut" oder mit Hilfe mathematischer Modelle analysiert werden. Dabei interessieren wir uns besonders für die Selbstorganisation biomolekularer und zellulärer Strukturen, die Optimierung der zugrundeliegenden aktiven und passiven Suchprozesse, sowie die sich daraus ergebende Informationsverarbeitung in biochemischen und neuronalen Netzwerken.
Biomedizinische Physik
Arbeitsgruppen: Hugel - Pfeiffer
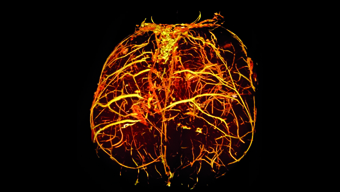 Biomedizinische bildgebende Verfahren. Bild:TUM.PH/Pfeiffer Lab. Die biomedizinische Physik erforscht die Anwendung von Prinzipien und Methoden der Grundlagenphysik auf medizinische Fragestellungen der vorklinischen oder klinischen Forschung. Im Fokus der Forschung stehen insbesondere neue bildgebende Methoden zur verbesserten Früherkennung und Diagnostik von Krankheiten, wie Krebs und Osteoporose, sowie deren Heilung durch den Einsatz neuartiger physikalischer Prinzipien. Ein weiteres Ziel ist ein molekulares Verständnis von Krankheiten, insbesondere solcher, die durch Amyloide verursacht werden (wie z.B. Parkinson oder Alzheimer). Die Erforschung, Entwicklung, und klinische Umsetzung neuartiger Röntgen-, Magnetresonanz-, oder Positronenemissions-Bildgebungsmethoden wird in enger Zusammenarbeit mit dem Zentralinstitut für Medizintechnik (IMETUM) und Medizinern am Klinikum rechts der Isar durchgeführt.
Biomedizinische bildgebende Verfahren. Bild:TUM.PH/Pfeiffer Lab. Die biomedizinische Physik erforscht die Anwendung von Prinzipien und Methoden der Grundlagenphysik auf medizinische Fragestellungen der vorklinischen oder klinischen Forschung. Im Fokus der Forschung stehen insbesondere neue bildgebende Methoden zur verbesserten Früherkennung und Diagnostik von Krankheiten, wie Krebs und Osteoporose, sowie deren Heilung durch den Einsatz neuartiger physikalischer Prinzipien. Ein weiteres Ziel ist ein molekulares Verständnis von Krankheiten, insbesondere solcher, die durch Amyloide verursacht werden (wie z.B. Parkinson oder Alzheimer). Die Erforschung, Entwicklung, und klinische Umsetzung neuartiger Röntgen-, Magnetresonanz-, oder Positronenemissions-Bildgebungsmethoden wird in enger Zusammenarbeit mit dem Zentralinstitut für Medizintechnik (IMETUM) und Medizinern am Klinikum rechts der Isar durchgeführt.