Wie regulieren Bakterien ihren Stoffwechsel?
Bakterieller Kontrollmechanismus zur Anpassung an wechselnde Umgebungsbedingungen
2017-11-29 – Nachrichten aus dem Physik-Department

Auf der Erde fluktuieren die Umweltbedingungen wie Temperatur, Licht, Verfügbarkeit von Nahrung und viele andere Parameter dauernd und ändern sich auch von Ort zu Ort. Das Leben, wie wir es kennen, ist nur möglich, weil Lebewesen die Fähigkeit haben, mit vielen unterschiedlichen Fluktuationen umzugehen. Jede Zelle und jeder Organismus hat unzählige Mechanismen, um sich geänderten Umweltbedingungen anzupassen.
Professor Ulrich Gerland erklärt: “Dies betrifft insbesondere die Bakterien, da sie ihre Umwelt nicht in ihrem Sinne beeinflussen können und auch nicht über größere Entfernungen wandern können. Sie müssen mehr oder weniger mit der Umgebung zurecht kommen, die sie vorfinden.”
Eines der am besten untersuchten Beispiele ist Escherichia Coli, ein Bakterium, das im Darm von Säugetieren, also auch Menschen, lebt. Dieses muss sich also permanent an die unterschiedliche Nahrung anpassen, die diese Tiere zu sich nehmen. Einige Proteine, wie Enzyme zum Spalten des Milchzuckers Laktose, sind nur notwendig, wenn Laktose tatsächlich vorhanden ist; andere Enzyme, die beispielsweise helfen bestimmte Aminosäuren zu synthetisieren, werden nur benötigt, wenn diese Aminosäuren fehlen.
Escherichia Coli passen sich an unterschiedliches Nahrungsangebot an
Im Labor untersuchen Wissenschaftler diese Anpassungen der Mikroorganismen, indem sie die Nahrung der Bakterien plötzlich ändern, also die Zusammensetzung der Nährlösung, in der sie gedeihen. Es ist allgemein akzeptiert und brachte Jacques Monod 1965 einen Nobelpreis, dass sich Bakterien anpassen, indem sie die Expression (also die Umsetzung der genetischen Information in Proteine) von bestimmten Proteinen regulieren. Je nach aktuellen Umweltbedingungen werden so die Konzentrationen unterschiedlicher Proteine angepasst.
Allerdings ist es trotz großen Interesses und gewaltiger Forschungsanstrengungen über mehr als ein halbes Jahrhundert nicht gelungen, die biochemischen Details dieses komplizierten Regelmechanismus vollständig aufzuklären und damit zu verstehen. Das ist auch nicht überraschend, wenn man berücksichtigt, dass selbst in einem eher “einfachen” Bakterium wie E. Coli etliche tausend unterschiedliche Proteine und andere Moleküle (in jeweils unterschiedlicher Konzentration) in der mikroskopisch winzigen Zelle dicht gepackt untergebracht sind. Hinzu kommt die entsprechende DNA, die Tausende von Genen enthält.
“Auch heute ist es nicht möglich, in Modellen alle Wechselwirkungen und biochemischen Reaktionen, die in einem Bakterium ablaufen, zu berücksichtigen,” erklärt Ulrich Gerland. “Aber alle Biomoleküle, die sich in einer Zelle befinden und alle Reaktionen, die ablaufen, sind Bestandteil des Stoffwechsels.” Bakterielles Wachstum durch Zellteilung bedeutet letztlich, dass das vollständige Repertoire an Biomolekülen innerhalb der Zelle von Grund auf verdoppelt wird.
Kinetik der Anpassung
Sein Team hat sich mit Kollegen von der UCSD auf die grundsätzlichen Mechanismen der Regulierung konzentriert und weniger auf die molekularen Details der Reaktionsketten. Die grundsätzliche Frage, die sich die Forscher experimentell stellten ist: Wie schnell passen sich Bakterien unterschiedlichen Änderungen ihrer Umgebung an?
Sie führten eine umfangreiche Studie durch, bei der die Wachstumsbedingungen der Bakterien geändert wurden. Zum Beispiel, indem nach einer schlechten Nahrungsquelle plötzlich reichlich Nahrung zur Verfügung gestellt wurde oder umgekehrt. Bei sogenannten Up-Shifts von Nahrungsstoffen erhöht sich die Wachstumsrate nach einer Verzögerung, die auf dem Anpassungsprozess beruht.
Andere Experimente führten zu einer sogenannten Diauxie, ein Phänomen das bereits Monod untersuchte. Wenn Bakterien mit einer bestimmten Sorte Nahrung versorgt werden und später mit anderen Nährstoffen, kommt es zeitweise zu einer Verlangsamung des Wachstums, obwohl ja immer ausreichend Nahrung vorhanden ist. Diese Verlangsamung beruht darauf, dass die Bakterien dann ihre Verdauung umstellen. Sie passen also die Konzentration von bestimmten Enzymen an – und deren Synthese dauert eine gewisse Zeit.
Während der Entwicklung der Bakterienkulturen wurde eine Reihe von Parametern erhoben, die eine genaue quantitative Charakterisierung der zeitlichen Entwicklung der Anpassung erlauben. Wie erwartet zeigten die unterschiedlichen Experimente qualitativ wie quantitativ unterschiedliche Wachstumskurven. Die Kurven hängen sowohl von den Anfangswerten als auch von den jeweiligen Änderungen ab.
Das Modell des Fließgleichgewichts
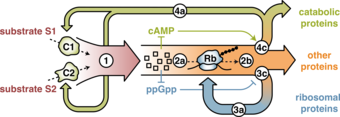
Die Forscher beließen es nicht bei diesen Beobachtungen. Um die Mechanismen der Anpassung genauer aufzuklären, entwickelten sie ein physikalisch motiviertes Modell. In einem Top-down-Ansatz verwendet das Modell lediglich qualitatives Wissen über die biochemischen Details der Regulationsmechanismen. Es basiert darauf, den Stofffluss in der Zelle zu bilanzieren, d.h. Gleichungen für den Materialtransport aufzustellen.
Es wurden unterschiedliche Regelmechanismen berücksichtigt, aber unter Berücksichtigung der Stoffbilanz können diese zu einer globalen Differenzialgleichung zusammengefasst werden. Dies führt zu einer globalen Betrachtung der Kinetiken der gesamten biochemischen Reaktionen, die bei den verschiedenen Anpassungen beteiligt sind.
“Unser Modell des Fließgleichgewichts der Regulierung beschreibt die zeitlichen Verläufe der Anpassungsprozesse an sich ändernde Nahrungsangebote wie Erhöhung, Senkung oder Wechsel des Nährstoffangebots quantitativ und ohne anpassbare Parameter richtig”, fasst Ulrich Gerland die Ergebnisse der Studie zusammen.
“Offenbar hängt die Kinetik der Wachstumsanpassung gar nicht von mikroskopischen Details der einzelnen biochemischen Reaktionen ab, sondern folgt einer globalen Strategie für die Umverteilung von Ressourcen für die Proteinsynthese”, sagt Ulrich Gerland. “Damit könnte unser theoretisches Modell auch für eine Reihe von ähnlichen kinetischen Vorgängen anwendbar sein.”
Unterstützt wurde die Forschung vom National Institutes of Health (NIH), USA, von der Simons Foundation, USA und von der Deutschen Forschungsgemeinschaft über das Exzellenzcluster ‘Nanosystems Initiative Munich’ sowie das Schwerpunktprogramm SPP1617.
- Redaktion
- Dr. Johannes Wiedersich
Veröffentlichungen
- A global resource allocation strategy governs growth transition kinetics of Escherichia coliDavid W. Erickson, Severin J. Schink, Vadim Patsalo, James R. Williamson, Ulrich Gerland & Terence HwaDOI: 10.1038/nature24299
- Quantifying the benefit of a proteome reserve in fluctuating environmentsMatteo Mori, Severin Schink, David W. Erickson, Ulrich Gerland, and Terence Hwa