DNA-Origami überwindet wichtige Grenzen
Aufbau von Strukturen in Virengröße und Kostensenkung durch Massenproduktion
2017-12-07 – Nachrichten aus dem Physik-Department

Viren schließen ihre Erbsubstanz in eine Kapsel ein, die aus vielen gleichen Proteinbausteinen besteht. Die Kapsel des Hepatitis-B-Virus beispielsweise besteht aus 180 identischen Untereinheiten, ein typisches Beispiel für die auch von der Natur gerne genutzte „Fertigteil-Bauweise“.
Das Team von Hendrik Dietz, Professor für Biomolekulare Nanotechnologie an der TU München, hat nun virale Konstruktionsprinzipien in die DNA-Origami-Technologie übertragen. Damit sind sie nun in der Lage, Strukturen von der Größe von Viren und Zellorganellen gezielt zu konstruieren und aufzubauen.
Grundlage der Technologie ist ein langer Einzelstrang, der durch kurze Gegenstücke zu einer doppelsträngigen Struktur ergänzt wird. „Die doppelsträngige Struktur ist energetisch so stabil, dass wir den Einzelstrang durch geschickt gewählte kurze Gegenstücke fast in beliebige Formen zwingen können“, erläutert Hendrik Dietz. „Mittlerweile können wir damit am Computer präzise Objekte konstruieren, die nur wenige Nanometer groß sind.“
Zahnräder für den Nanomotor
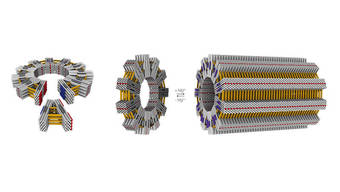
Mittlerweile beherrscht die Arbeitsgruppe auch Techniken, die Objekte durch hinzugefügte Seitengruppen weiter zu modifizieren. Doch die Größe der Objekte blieb auf die Nanometerskala beschränkt. In ihrer im renommierten Fachjournal „Nature“ erschienenen Publikation zeigen sie nun, wie man aus Fertigbauteilen größere Strukturen aufbauen kann.
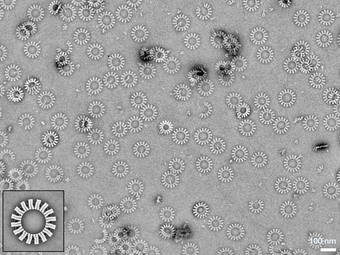
Dazu schufen sie zunächst Nanoobjekte in V-Form. Diese bekamen an den Seiten form-komplementäre Bindungsstellen, so dass sie sich in der Lösung schwimmend von selbst zusammenfügten. Je nach Öffnungswinkel entstanden so „Zahnräder“ aus zehn bis 28 Einzelelementen.
„Zu unserer großen Freude entstehen fast ausschließlich die durch den Öffnungswinkel der Einzelelemente vorgegebenen Ringe“, sagt Hendrik Dietz. „Entscheidend dafür, dass wir überhaupt so groß und komplex bauen können, sind die Präzision und die Steifigkeit der einzelnen Bausteine. Die Einzelelemente mussten wir beispielsweise mit Querstreben versteifen.“
Aufbau von Mikroröhrchen
Um das Strukturprinzip weiter auszureizen schuf das Team neue Moleküle, die nicht nur „Klebestellen“ an der Seite, sondern zusätzlich etwas schwächere Verbindungsstellen auf Ober- und Unterseite besitzen. Nun setzen sich die „Nanozahnräder“ in einem zweiten Schritt über die zusätzlichen Bindungsstellen zu langen Röhrchen zusammen.
„Mit Längen von einem Mikrometer und Durchmessern von mehreren Hundert Nanometern erreichen diese Röhrchen bereits die Größe von Stäbchenbakterien“, erläutert Hendrik Dietz. „Und über die Architektur der Einzelbausteine können wir die Gesamtstruktur präzise bestimmen.“
Aufbau polyedrischer Strukturen
Inspiriert von den Symmetrien und dem hierarchischen Aufbau von Viren, versuchten die Forscher auch Käfigstrukturen aufzubauen. „Eine mögliche zukünftige Anwendung künstlicher Käfige könnte der Transport von Medikamenten im Körper sein“, erläutert Hendrik Dietz die Motivation. „Das Ziel dabei ist, die Wirkstoffe ausschließlich am Einsatzort freizusetzen und den restlichen Körper zu schonen.“
Nach den bereits bei den vorherigen Strukturen angewandten Prinzipien konstruierte das Team nun Bauteile, die sich unter den richtigen Bedingungen zu Käfigstrukturen zusammenfügen sollten. Aus einem dreieckigen Mittelstück und drei V-förmigen Bausteinen entsteht so wieder ein dreiarmiges Bauteil.
Je nach Öffnungswinkel des V fügen sich mehrere davon im zweiten Schritt zu tetraedrischen, hexaedrischen oder dodekaedrischen Strukturen zusammen. Diese Strukturen integrieren dabei bis zu 1.8 Millionen addressierbare DNA Basenpaare an definierten Positionen. Die Käfige erreichen damit erstmals die Größe von Viren und kleinen Zellorganellen.
Kostengünstige Massenproduktion

Bisher beschränken die Herstellungsverfahren die Einsatzmöglichkeiten auf Felder, bei denen nur geringe Mengen benötigt werden. Das mit den derzeit gängigen Methoden nur wenige Mikrogramm hergestellt werden können, schließt denkbare Anwendungen in der Medizin oder in der Materialwissenschaft völlig aus.
Das Nadelöhr sind dabei die kurzen Klammerstränge, die aufwändig Base für Base chemisch aufgebaut werden müssen. Der aus Bakteriophagen gewonnene Hauptstrang dagegen kann einfach und in großen Mengen biotechnologisch produziert werden.
Das Team um Hendrik Dietz entwickelte daher eine aus der synthetischen Biotechnologie stammende Entdeckung weiter, sogenannte DNA-Enzyme. Dies sind DNA-Stücke, die bei einer hohen Konzentration von Zink-Ionen an bestimmten Stellen auseinanderbrechen.
Die kurzen Klammer-Sequenzen verbanden sie mit jeweils zwei modifizierten DNA-Enzymen zu einem langen Strang. „Wie der Einzelstrang einer Bakteriophagen-DNA lässt sich ein solcher Strang, einmal präzise mit der richtigen Basenfolge hergestellt, mit biotechnologischen Verfahren vervielfältigen“, erläutert Dietz den Trick des Verfahrens.
Biotechnologische Produktion im großen Maßstab
Die Herstellung sowohl des Hauptstrangs als auch des aus DNA-Enzymen und Klammer-Sequenzen bestehenden zweiten Strangs gelang in einem Hochzelldichte-Verfahren mit Bakterien. Da dieses skalierbar ist, sind Hauptstrang und Klammern auch in großen Mengen produzierbar. Erhöht man nach Isolierung der DNA die Konzentration an Zink-Ionen, werden die kurzen Klammerstränge freigesetzt und können den Hauptstrang in die gewünschte Form falten.
Umfangreiche reaktionstechnische Untersuchungen zusammen mit Kollegen am Lehrstuhl für Bioverfahrenstechnik zeigten, dass dies auch in großem Volumen möglich ist. Im Technikum für Weiße Biotechnologie der TU München in Garching konnten die Wissenschaftler so bereits Grammmengen von vier verschiedenen DNA-Origami-Objekten herstellen. Auch die weitere Skalierbarkeit in den Kubikmeter-Maßstab erscheint nun möglich.
„Im Zusammenspiel von Biotechnologie und Verfahrenstechnik ist damit ein wirklich grundlegender Meilenstein auf dem Weg für zukünftige Anwendungen der DNA-Nanotechnologie gelungen“, sagt Mitautor Professor Dirk Weuster-Botz, Inhaber des Lehrstuhls für Bioverfahrenstechnik.
Die Arbeiten wurden unterstützt mit Mitteln des European Research Council, der Deutschen Forschungsgemeinschaft (DFG) aus dem Gottfried-Wilhelm-Leibniz Programm, dem SFB 863, den Exzellenzclustern Center for Integrated Protein Science Munich (CIPSM) und Nanosystems Initiative Munich (NIM), der International Graduate School of Science and Engineering, dem aus Mitteln der DFG und der Europäischen Gemeinschaft geförderten Institute for Advanced Study der Technischen Universität München, des BioOrigami-Projekts des Bundesministeriums für Bildung und Forschung (BMBF) und der Bosch Forschungsstiftung.
- Redaktion
- Andreas Battenberg, Dr. Johannes Wiedersich
Veröffentlichungen
- Gigadalton-scale shape-programmable DNA assembliesKlaus F. Wagenbauer, Christian Sigl und Hendrik DietzDOI: 10.1038/nature24651
- Biotechnological mass production of DNA origamiFlorian Praetorius, Benjamin Kick, Karl L. Behler, Maximilian N. Honemann, Dirk Weuster-Botz und Hendrik DietzDOI: 10.1038/nature24650
Verwandte Meldungen
- Designer-Proteine falten DNA – 2017-03-24
- Auf der molekularen Streckbank – 2017-02-24
- Zwei weitere ERC-Grants ans Physik-Department – 2016-12-23
- Kräftemessen im Erbgutmolekül – 2016-09-12
- Biophysiker Hendrik Dietz (36) erhält deutsche Spitzenauszeichnung – 2014-12-10